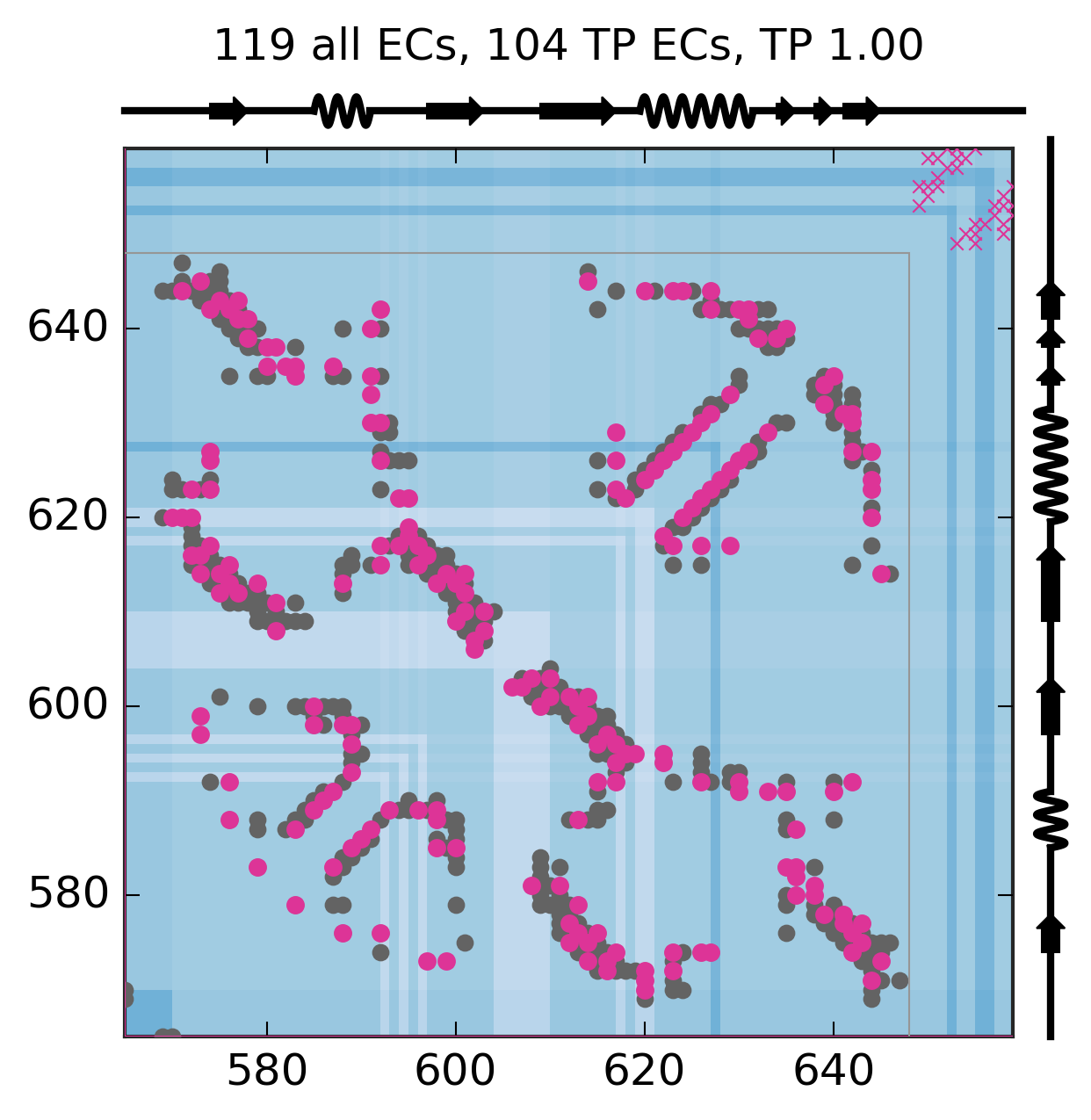
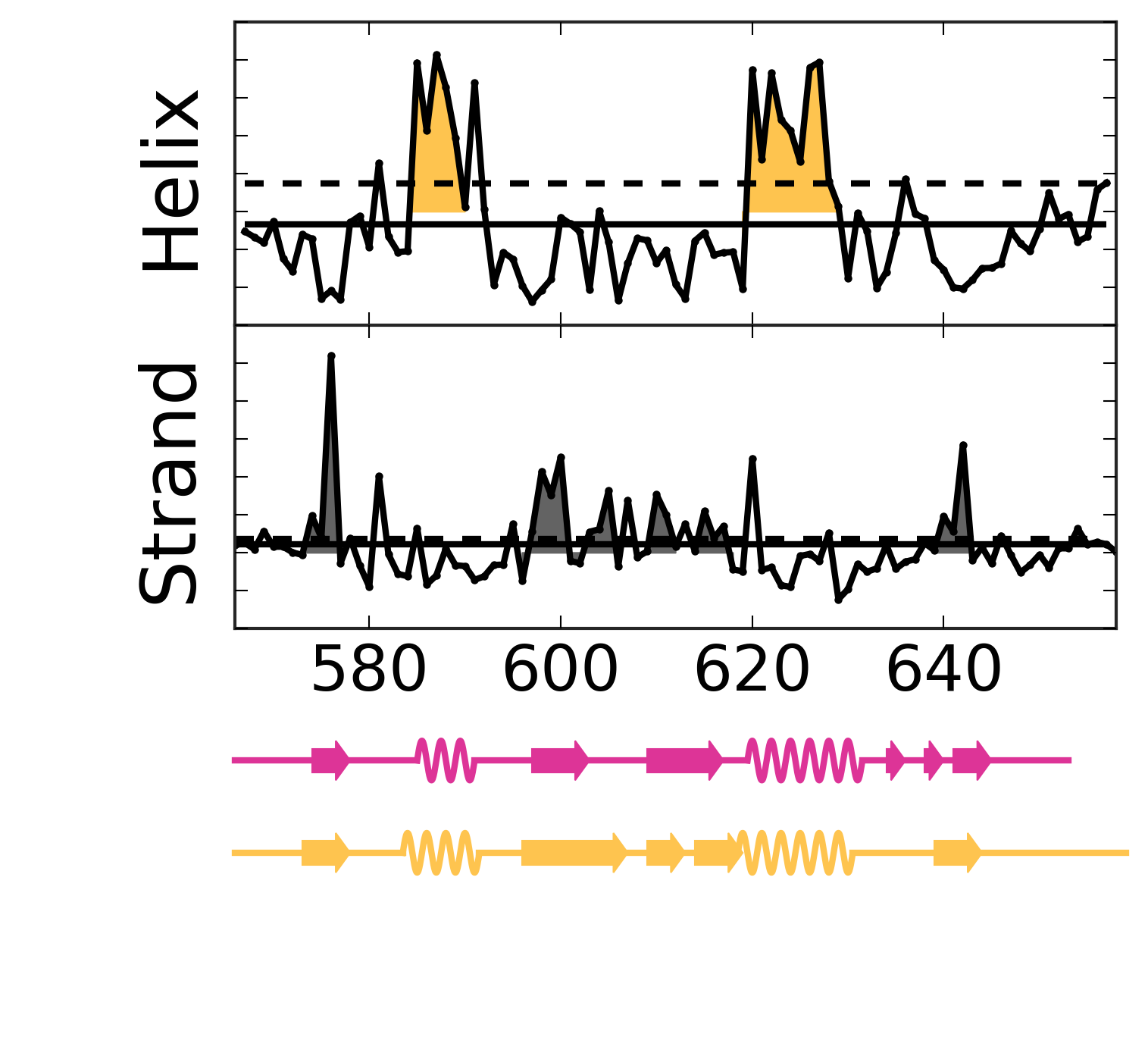
Evolutionary Coupling Analysis
Predicted and experimental contacts
Secondary structure from ECs
EC score distribution and threshold
Top ECs
| Rank |
Residue 1 |
Amino acid 1 |
Residue 2 |
Amino acid 2 |
EC score |
| 1 |
573 |
T |
616 |
D |
2.83 |
| 2 |
595 |
S |
618 |
N |
2.80 |
| 3 |
587 |
T |
636 |
D |
2.63 |
| 4 |
634 |
E |
639 |
K |
2.28 |
| 5 |
617 |
F |
626 |
A |
2.19 |
| 6 |
601 |
V |
612 |
F |
2.11 |
| 7 |
620 |
E |
624 |
K |
2.11 |
| 8 |
577 |
K |
641 |
T |
2.09 |
| 9 |
625 |
A |
629 |
A |
2.05 |
| 10 |
585 |
E |
589 |
K |
1.95 |
| 11 |
592 |
F |
615 |
V |
1.89 |
| 12 |
623 |
A |
644 |
W |
1.88 |
| 13 |
618 |
N |
622 |
D |
1.79 |
| 14 |
597 |
R |
616 |
D |
1.73 |
| 15 |
591 |
S |
633 |
G |
1.73 |
| 16 |
572 |
K |
623 |
A |
1.72 |
| 17 |
624 |
K |
644 |
W |
1.70 |
| 18 |
620 |
E |
644 |
W |
1.69 |
| 19 |
572 |
K |
620 |
E |
1.65 |
| 20 |
589 |
K |
593 |
D |
1.63 |
| 21 |
627 |
K |
644 |
W |
1.61 |
| 22 |
591 |
S |
635 |
I |
1.61 |
| 23 |
583 |
T |
587 |
T |
1.61 |
| 24 |
581 |
E |
638 |
N |
1.60 |
| 25 |
635 |
I |
640 |
V |
1.60 |
| 26 |
574 |
L |
627 |
K |
1.59 |
| 27 |
601 |
V |
614 |
F |
1.59 |
| 28 |
574 |
L |
623 |
A |
1.58 |
| 29 |
632 |
D |
639 |
K |
1.56 |
| 30 |
600 |
I |
609 |
S |
1.54 |
| 31 |
595 |
S |
622 |
D |
1.53 |
| 32 |
577 |
K |
643 |
D |
1.51 |
| 33 |
579 |
L |
613 |
G |
1.50 |
| 34 |
576 |
V |
592 |
F |
1.50 |
| 35 |
624 |
K |
628 |
E |
1.42 |
| 36 |
591 |
S |
630 |
M |
1.39 |
| 37 |
583 |
T |
635 |
I |
1.34 |
| 38 |
577 |
K |
612 |
F |
1.32 |
| 39 |
578 |
G |
641 |
T |
1.31 |
| 40 |
575 |
F |
612 |
F |
1.30 |
| 41 |
588 |
L |
613 |
G |
1.26 |
| 42 |
572 |
K |
616 |
D |
1.26 |
| 43 |
580 |
S |
636 |
D |
1.26 |
| 44 |
600 |
I |
613 |
G |
1.25 |
| 45 |
581 |
E |
608 |
S |
1.25 |
| 46 |
622 |
D |
626 |
A |
1.25 |
| 47 |
582 |
D |
636 |
D |
1.23 |
| 48 |
594 |
G |
617 |
F |
1.23 |
| 49 |
586 |
E |
590 |
E |
1.22 |
| 50 |
621 |
E |
625 |
A |
1.21 |
| 51 |
592 |
F |
626 |
A |
1.19 |
| 52 |
580 |
S |
638 |
N |
1.18 |
| 53 |
589 |
K |
598 |
A |
1.16 |
| 54 |
571 |
S |
644 |
W |
1.14 |
| 55 |
654 |
G |
658 |
G |
1.13 |
| 56 |
576 |
V |
588 |
L |
1.09 |
| 57 |
574 |
L |
626 |
A |
1.09 |
| 58 |
655 |
G |
659 |
G |
1.07 |
| 59 |
585 |
E |
598 |
A |
1.05 |
| 60 |
581 |
E |
611 |
G |
1.04 |
| 61 |
627 |
K |
642 |
L |
1.03 |
| 62 |
592 |
F |
630 |
M |
1.03 |
| 63 |
598 |
A |
613 |
G |
1.02 |
| 64 |
585 |
E |
600 |
I |
1.01 |
| 65 |
591 |
S |
640 |
V |
1.01 |
| 66 |
576 |
V |
615 |
V |
1.01 |
| 67 |
592 |
F |
642 |
L |
1.00 |
| 68 |
631 |
E |
641 |
T |
0.99 |
| 69 |
571 |
S |
620 |
E |
0.99 |
| 70 |
583 |
T |
636 |
D |
0.98 |
| 71 |
573 |
T |
645 |
A |
0.96 |
| 72 |
626 |
A |
630 |
M |
0.96 |
| 73 |
630 |
M |
642 |
L |
0.96 |
| 74 |
576 |
V |
642 |
L |
0.93 |
| 75 |
651 |
G |
655 |
G |
0.92 |
| 76 |
573 |
T |
597 |
R |
0.91 |
| 77 |
574 |
L |
617 |
F |
0.91 |
| 78 |
587 |
T |
591 |
S |
0.90 |
| 79 |
573 |
T |
599 |
R |
0.88 |
| 80 |
596 |
V |
615 |
V |
0.87 |
| 81 |
653 |
F |
658 |
G |
0.85 |
| 82 |
602 |
T |
607 |
G |
0.85 |
| 83 |
579 |
L |
583 |
T |
0.85 |
| 84 |
603 |
D |
608 |
S |
0.84 |
| 85 |
602 |
T |
606 |
T |
0.84 |
| 86 |
614 |
F |
645 |
A |
0.83 |
| 87 |
627 |
K |
631 |
E |
0.83 |
| 88 |
575 |
F |
614 |
F |
0.82 |
| 89 |
653 |
F |
659 |
G |
0.81 |
| 90 |
650 |
E |
658 |
G |
0.81 |
| 91 |
570 |
P |
620 |
E |
0.80 |
| 92 |
651 |
G |
656 |
R |
0.79 |
| 93 |
653 |
F |
657 |
G |
0.79 |
| 94 |
588 |
L |
598 |
A |
0.78 |
| 95 |
649 |
G |
653 |
F |
0.78 |
| 96 |
649 |
G |
655 |
G |
0.75 |
| 97 |
575 |
F |
643 |
D |
0.75 |
| 98 |
652 |
G |
657 |
G |
0.74 |
| 99 |
603 |
D |
610 |
K |
0.73 |
| 100 |
650 |
E |
654 |
G |
0.73 |
| 101 |
596 |
V |
617 |
F |
0.73 |
| 102 |
592 |
F |
617 |
F |
0.73 |
| 103 |
650 |
E |
655 |
G |
0.73 |
| 104 |
589 |
K |
596 |
V |
0.71 |
| 105 |
623 |
A |
627 |
K |
0.71 |
| 106 |
594 |
G |
622 |
D |
0.70 |
| 107 |
651 |
G |
658 |
G |
0.70 |
| 108 |
573 |
T |
614 |
F |
0.69 |
| 109 |
601 |
V |
610 |
K |
0.69 |
| 110 |
578 |
G |
639 |
K |
0.69 |
| 111 |
599 |
R |
614 |
F |
0.69 |
| 112 |
617 |
F |
629 |
A |
0.69 |
| 113 |
574 |
L |
642 |
L |
0.68 |
| 114 |
617 |
F |
623 |
A |
0.68 |
| 115 |
595 |
S |
619 |
S |
0.67 |
| 116 |
631 |
E |
642 |
L |
0.67 |
| 117 |
652 |
G |
659 |
G |
0.67 |
| 118 |
576 |
V |
613 |
G |
0.66 |
| 119 |
629 |
A |
633 |
G |
0.66 |
Alignment robustness analysis
First most common residue correlation
Second most common residue correlation